和蛋白-DNA交联不同的是,这种树枝状高分子不会影响酶切,从而有效降低了染色质互作图上的背景。联合DNase酶切,提高染色质互作图的分辨率的同时也不带来任何偏好性。

然而利用甲醛交联DNA和蛋白的反应是可逆的,且交联产物并不稳定。此外,交联于DNA上的蛋白在空间上有概率阻挡限制性内切酶识别DNA,造成DNA不完全酶切。因此在不同细胞中连接位点也会不同,大大增加了异质性从而增加染色质互作图上的“背景”,影响染色质结构的正确解析。此外,相比于限制性内切酶,DNase和MNase能将DNA切成更小的片段,因此基于DNase和MNase发明的衍生技术DNase Hi-C和Micro-C能大大提高染色质互作图的分辨率【4】。然而由于依旧是依靠甲醛交联DNA和蛋白,DNase和MNase将更偏向于酶切染色质开放区域。因此观测到的染色体结构也会有偏好性。
该研究表明,相比于Hi-C,CAP-C能够捕获到完全相似的染色质结构(loop,stripe,TADs,compartment)。由于CAP-C染色质互作图“背景”更低,有更多的小于50Kb大小的染色质结构域被发现,同时也使得CAP-C能够检测到更多的染色质环。相比于此前人们发现的染色质结构域(例如TADs),这些小染色质结构域的边界(boundary)富集了更多的H3K4me3,H3K27ac组蛋白修饰。并且有部分基因独立形成了一个染色质结构域(图1)。

图1. CAP-C实验设计。CAP-C能有效提高染色质互作图上的“信噪比”,帮助发现更多染色质精细结构。
研究人员进一步将结构域分为环状(loop)结构域和非环状(non-loop)结构域,同过对比环状结构域,发现非环状结构域显著性的更小;有更多的H3K4me3,H3K27ac组蛋白修饰富集于非环状结构域的边界;非环状结构域里的基因转录水平更高。近年来有大量研究表明环状结构域可能是通过cohesin和CTCF形成的染色质环在DNA上滑动挤压形成的【5】。而鲜有研究证明非环状结构域的形成机制。研究人员发现敲除CTCF更显著的降低了环状结构域中的染色质互作而抑制转录则更显著的降低了非环状结构域中的染色质互作。因此研究人员推测形成这两种结构域的分子机制可能不同 (图2)。
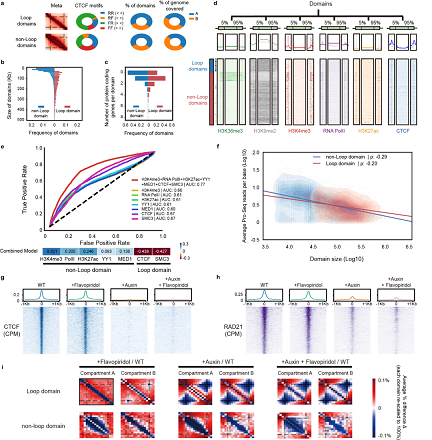
图2. 环状结构域和非环状结构域的对比
研究人员还进一步发现若基因内部存在活跃的可选择性启动子,会在该启动子的位置上形成结构域边界并将一个基因分割进多个结构域中。若抑制转录水平,这些边界将变得不再那么明显。通过比较这些启动子上的RNA聚合酶II和CTCF的水平发现,RNA聚合酶II及CTCF的结合并不是这些结构域边界的形成所必须的,该结果与之前的研究发现类似【6】。因此研究人员推测这些结构域边界可能有其他更多的活跃转录机器共同调控形成。此外,激活异染色质上的转录将在转录位置形成结构域边界,并出现B 区室向A区室的转换。同样的,结构域边界的形成及区室的转换与转录激活相关但与 RNA聚合酶II的结合无关。
研究人员还观察到当抑制转录后会增强部分区域染色质互作,这是之前从未报道过的现象。研究人员发现这些区域通常是由两个或多个高表达基因的启动子互作形成的。抑制转录后,这些启动子上的RNA聚合酶II水平显著的上升,由RNA聚合酶II介导的染色质环也显著的增加。通过抑制RNA聚合酶II与启动子的结合,这些区域将不再出现染色质互作。由于该结构的形成与转录起始及RNA聚合酶II与启动子的结合相关,因此研究人员将这些区域命名为转录起始簇(transcription initiation cluster,TIC)。此前有大量研究表明RNA聚合酶II在体内能够通过相分离形成聚合体【7】,因此研究人员猜测这种染色质结构的形成可能与相分离有关。具体的分子机制还有待更多的实验证据(图3)。
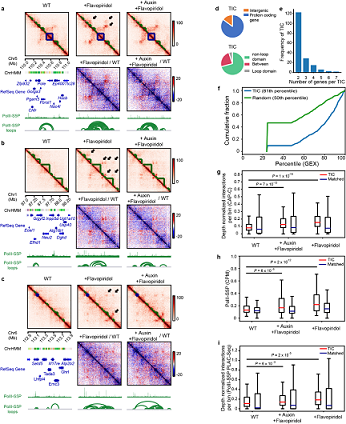
图3. 转录起始簇
由于树枝状高分子(dendrimer)是一个分子大小可调控的高分子,其大小是通过高分子合成代数(generation)控制的,代数越大分子越大,因此所修饰形成的交联平台也越大,理论上能够抓住空间上相距更远的DNA。研究人员通过比较不同大小的交联平台所捕获的染色质构象发现,小的树枝状高分子(如G3)捕获到更多B 区室间的染色质互作而大的树枝状高分子(如G5或者G7)捕获到更多A区室间的染色质互作。
由于B区室与异染色质相关,小的树枝状高分子更容易钻进缠绕紧密的异染色质之间;而A区室与常染色质相关,大的树枝状高分子通过碰撞能捕获到更多松散的常染色质结构。因此观测到的实验结果符合预期。研究人员进一步通过对大小不同的树枝状高分子所捕获的染色质互作区域进行主成分分析发现区室并不是只存在于低分变率下的染色质结构。同样在高分辨率下也存在。研究人员在老鼠,人和果蝇细胞中均发现了之前并未报道过的精细的区室结构的存在。这些不同大小的树枝状高分子交联平台在未来可能可以作为“分子尺“,测量并绘制染色体的三维结构(图4)。

图4. 精细的区室结构存在于各类物种中。图中,染色质被小树枝状高分子(G3)更多的捕获的区域用红色标注,染色质被大树枝状高分子(G5或G7)更多的捕获的区域用蓝色标注
总的来说,CAP-C发现了之前并未报道过的染色质精细结构(small non-loop domain,compartment及TIC),这些结构均与转录相关。具体的分子机制还有待更多的实验结果去证明阐释。究竟转录是否会影响染色质结构还是一个未解的难题,需要结合更多的生物学研究方法来论证。CAP-C作为新型的交联方法,可预见其应用前景十分广泛。由于树枝状高分子能够直接交联DNA,CAP-C理论上也能捕获天然染色质(native chromatin)的结构。
而天然染色质可能更能反映染色体在生理状态下的折叠情况。此外,CAP-C的交联方式可以与其他下游实验相结合,例如(GAM或SPRITE)【8,9】,从而可以通过多个角度来研究染色体结构。树枝状高分子上也可被修饰其他化学官能基团,使得CAP-C能够更广阔的应用于捕获其他大分子间的相互作用(RNA-RNA, RNA-DNA)。
据悉,何川课题组游乾承博士和谷茜博士以及欧阳正清课题组Anthony Cheng为本文的共同第一作者,何川教授为本课题设计提供了指导和支持,欧阳正清教授指导了数据挖掘和统计分析,任兵教授为数据解读提供了指导。
参考文献
1. Rao, S. S. P. et al. A 3D Map of the Human Genome at Kilobase Resolution Reveals Principles of Chromatin Looping. Cell 159, 1665–1680 (2014).
2. Dixon, J. R. et al. Topological domains in mammalian genomes identified by analysis of chromatin interactions. Nature 485, 376–380 (2012).
3. Lieberman-Aiden, E. et al. Comprehensive Mapping of Long-Range Interactions Reveals Folding Principles of the Human Genome. Science 326, 289–293 (2009).
4. Hsieh, T. H. S. et al. Resolving the 3D Landscape of Transcription-linked Mammalian Chromatin Folding. Mol. Cell 78, 539-553.e8 (2020).
5. Rao, S. S. P. et al. Cohesin Loss Eliminates All Loop Domains. Cell 171, 305-320.e24 (2017).
6. Bonev, B. et al. Multiscale 3D Genome Rewiring during Mouse Neural Development. Cell 171, 557-572.e24 (2017).
7. Lu, H. et al. Phase-separation mechanism for C-terminal hyperphosphorylation of RNA polymerase II. Nature 558, 318–323 (2018).
8. Beagrie, R. A. et al. Complex multi-enhancer contacts captured by genome architecture mapping. Nature 543, 519–524 (2017).
9. Quinodoz, S. A. et al. Higher-Order Inter-chromosomal Hubs Shape 3D Genome Organization in the Nucleus. Cell 174, 744-757.e24 (2018).
文章来源: BioArt