
来源:medRxiv
首都医科大学陈德喜教授团队基于illumina Nextera XT Tn5转座酶直接切割RNA/DNA杂合链的特性[5],优化mNGS建库方法,并在NextSeq 500平台上对该方法在临床检测呼吸道RNA病毒的各项性能指标进行了确认,包括低检测限、精密度、稳定性、抗干扰能力以及SARS-CoV-2病毒检测准确性等,为mNGS临床实验室常规检测应用提供了数据参考和支持。5月14日,该研究成果发表在预印本medRxiv上[6]。
一般mNGS的RNA检测流程包括逆转录、二链cDNA合成、预扩增/等温扩增、cDNA片段化和PCR扩增等流程[7]。研究团队通过优化RNA建库步骤、缩短建库时间,使mNGS操作简便,更适合临床使用。该团队将这种简便、快速、高灵敏度的检测方法命名为CATCH(pathogeniCrnA/dna hybrid Tagmentation teCHnology)。CATCH可跳过二链cDNA合成,通过优化延伸和PCR步骤,将整个文库制备过程缩短为3小时,人工处理时间为35分钟(图1)。该方法对SARS-CoV-2的检测限为39.2拷贝/test,A型流感病毒为278.1拷贝/mL,其重复性、稳定性和抗干扰性都表现出了较好的性能(表1)。
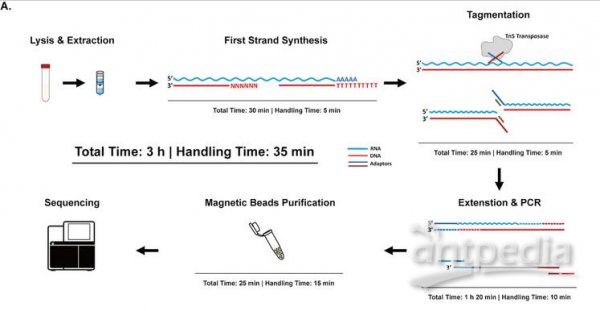
图1. CATCH工作流程示意图。来源:medRxiv

表1. CATCH对A型流感病毒和SARS-CoV-2的检测性能特征。来源:medRxiv
该方法理论上可以多重检测所有RNA病毒,研究团队选取了7种常见呼吸道RNA病毒,在接近检测下限处设置病毒载量基线,基于健康人咽拭子VTM基质模拟7种病毒混合样本,验证了该方法的鉴别检测能力。结果发现,CATCH不仅可以精确的鉴别7种病毒,并能对A型流感病毒进行亚型区分(图2),研究还发现mNGS检测信号与病毒浓度具有显著相关性,说明该方法还可以达到半定量的效果(图3)。
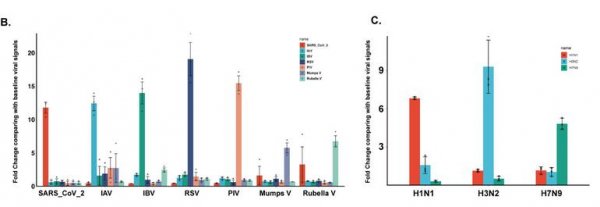
图2.CATCH在RNA病毒检测中的应用。(B)7种典型呼吸道RNA病毒:A型流感病毒(A / 2009 / H1N1)、B型流感病毒(B / Vitoria)、呼吸道合胞病毒、副流感病毒、腮腺炎病毒和风疹病毒的鉴别检测和半定量。(C)3种典型的A型流感病毒亚型的鉴别检测和半定量。来源:medRxiv
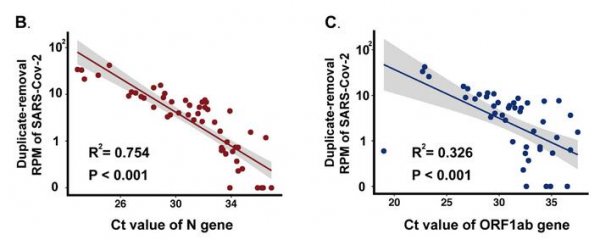
图3.(B)qRT-PCR ORF1ab基因Ct值和mNGS检测病毒信号的比较。(C)qRT-PCR N基因Ct值和mNGS病毒信号的比较。来源:medRxiv
研究团队回顾性的纳入了北京佑安医院疫情期间的64位COVID-19患者的98个RNA样本,同时采用CATCH和qRT-PCR两种方法检测。在47个双基因阳性的qPCR结果中,44个mNGS检测阳性的样本中存在SARS-CoV-2信号。在11个单基因阳性样本中,5个样本中可检测到SARS-CoV-2。此外,在40个qPCR检测为阴性的样本中,mNGS检测结果仍为阴性。在这些样本中,没有其他DNA或RNA病毒被检测为阳性。
总体而言,与qRT-PCR临床检测结果相比,CATCH的总体准确度为91.4%,灵敏度为84.5%,特异性为100%。如果仅考虑双基因阳性样品,其灵敏性将提高到93.7%(图4)。由于各种原因,临床样本的核酸浓度成为制约mNGS的因素之一。研究团队发现有相当一部分样本提取RNA低于qubit检测下限(65%,38.5%),无法满足以往的mNGS建库方法需要的起始量。CATCH方法可以超低起始量建库,对于低于qubit检测下限的RNA建库样本,成功率仍为100%。
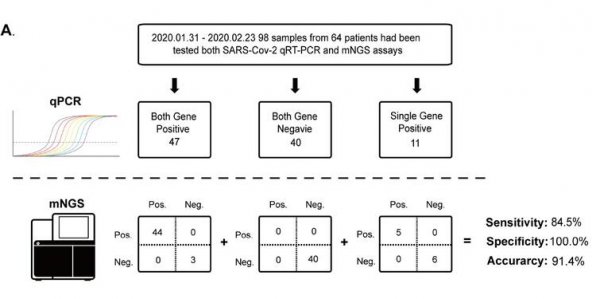
图4. CATCH与qRT-PCR检测SARS-CoV-2的性能比较。来源:medRxiv
据悉,该研究为首次对基于Tn5转座酶RNA快速建库mNGS方法进行的分析性能确认。准确可靠的临床性能验证是mNGS作为常规诊断检测推出之前必不可少的环节。该团队期待其研究工作能够为同行提供mNGS在临床开展所需要的具体方案和详实的数据,为mNGS病原检测真正走进临床助力。
参考文献:
[1] Zhu N, Zhang DY, Wang WL, et al. A Novel Coronavirus from Patients withPneumonia in China, 2019. N Engl J Med 2020; 382:727-33.
[2] Wu F, Zhao S, Yu B, et al. A new coronavirus associated with human respiratorydisease in China. Nature 2020; 579:265-+.
[3] 宋振举等.宏基因组分析和诊断技术在急危重症感染应用专家共识组.宏基因组分析和诊断技术在急危重症感染应用的专家共识[J].中华急诊医学杂志,2019, 028(002):151-155.
[4] Han D, Li Z, Li R, et al. mNGS in clinical microbiology laboratories: on theroad to maturity[J]. Critical Reviews in Microbiology, 2019(3):1-18.
[5] Di L, Fu Y, Sun Y, et al. RNA sequencing by direct tagmentation of RNA/DNAhybrids. Proc Natl Acad Sci U S A. 2020;117(6):2886‐2893.
[6] Laboratory validation of an RNA/DNA hybrid tagmentation based mNGS workflow on SARS-CoV-2 and other respiratory RNA viruses detection, medRxiv, May 14, 2020. doi: https://doi.org/10.1101/2020.05.12.20099754
[7] Miller S, Naccache SN, Samayoa E, et al. Laboratory validation of a clinicalmetagenomic sequencing assay for pathogen detection in cerebrospinal fluid.Genome Res 2019; 29: 831-42.